Susceptibilidad de la variante ómicron a los anticuerpos monoclonales y otros antivirales
Las mutaciones en la proteína Spike de ómicron hacen que, in vitro, esta variante sea resistente a la práctica totalidad de los anticuerpos monoclonales (Tabla 1).
En cambio, es esperable que ómicron siga siendo susceptible a antivirales de molécula pequeña como el inhibidor de proteasa CL3, nirmatrelvir (Paxlovid®, Pfizer), el análogo a ribonucleósidos, molnupiravir (MSD), y el inhibidor de la polimerasa, remdesivir (Veklury®, Gilead Sciences).
Tabla 1
Susceptibilidad in vitro de las variantes virales a los anticuerpos monoclonales.
Veces que se reduce la susceptibilidad a la neutralización (fold change)
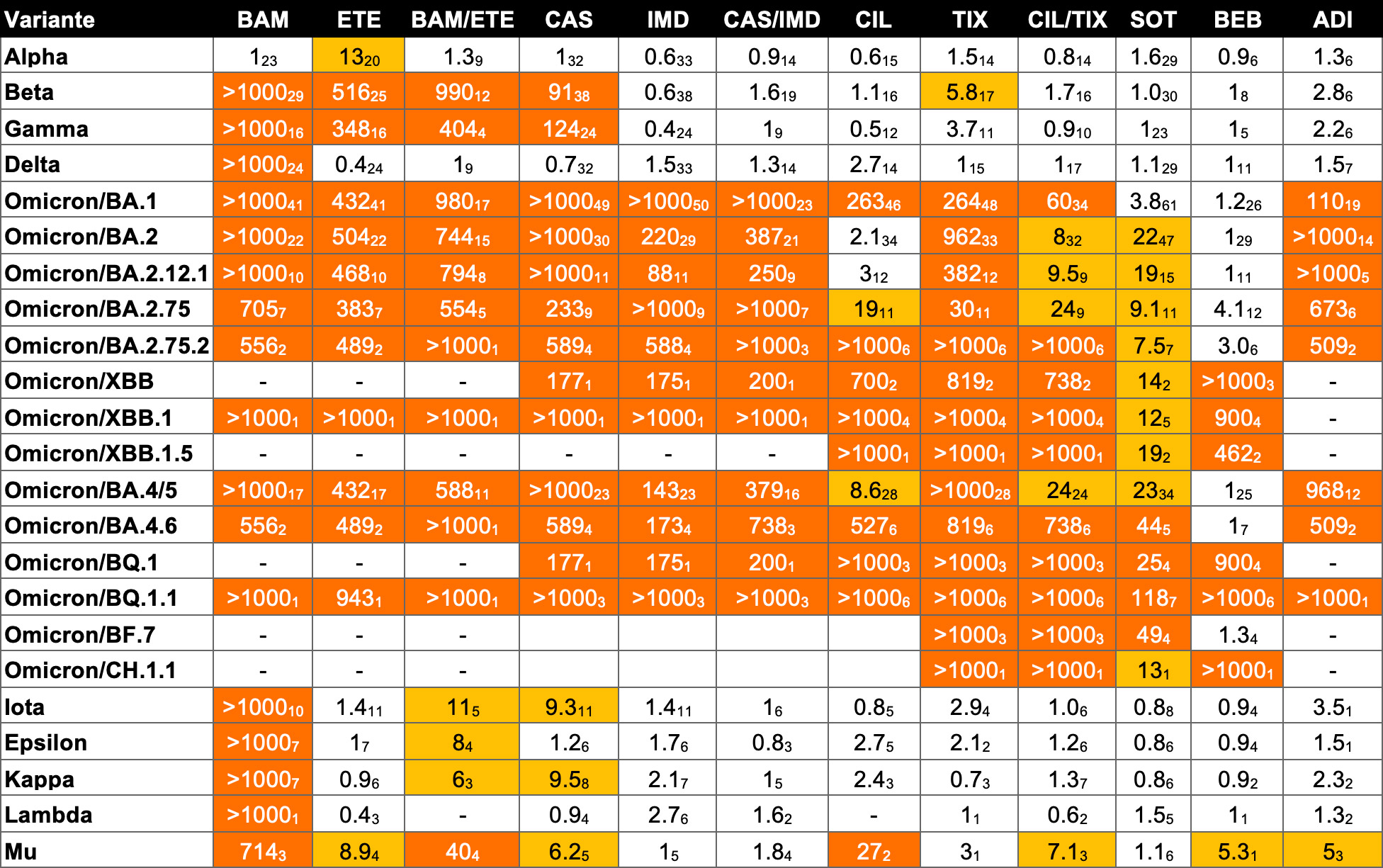
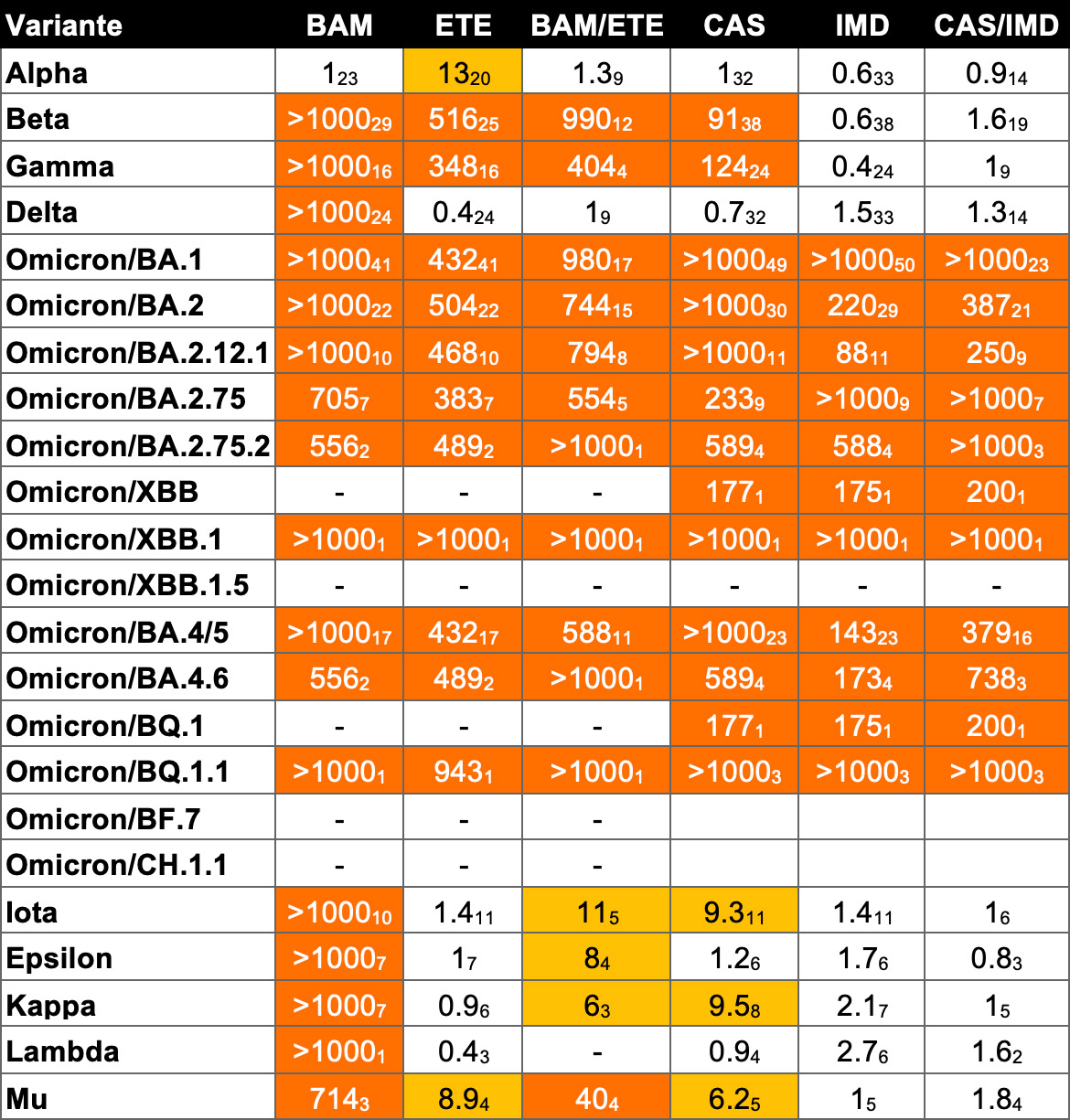
Tabla 1 (continuación)
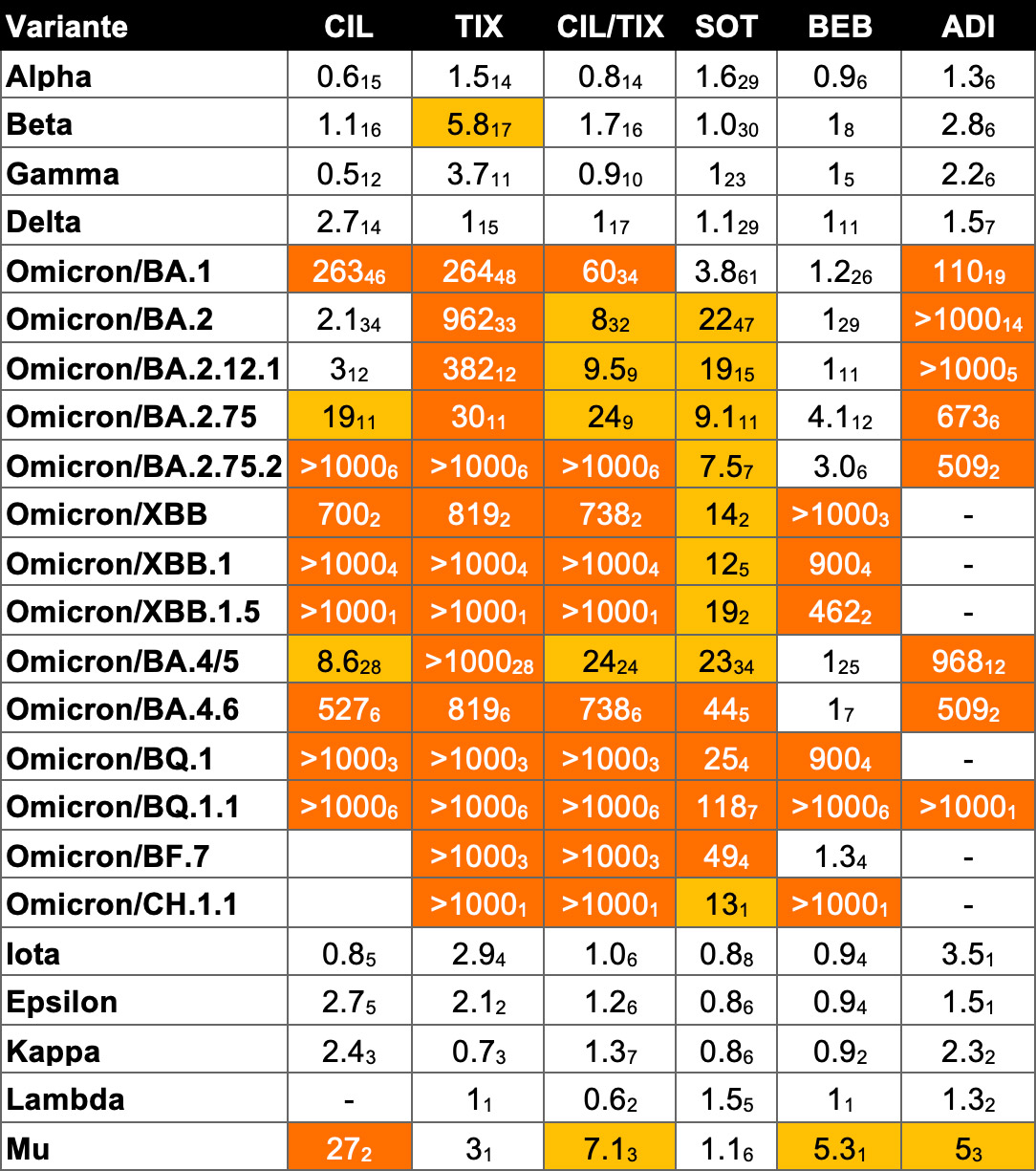
Tabla 1 (continuación)
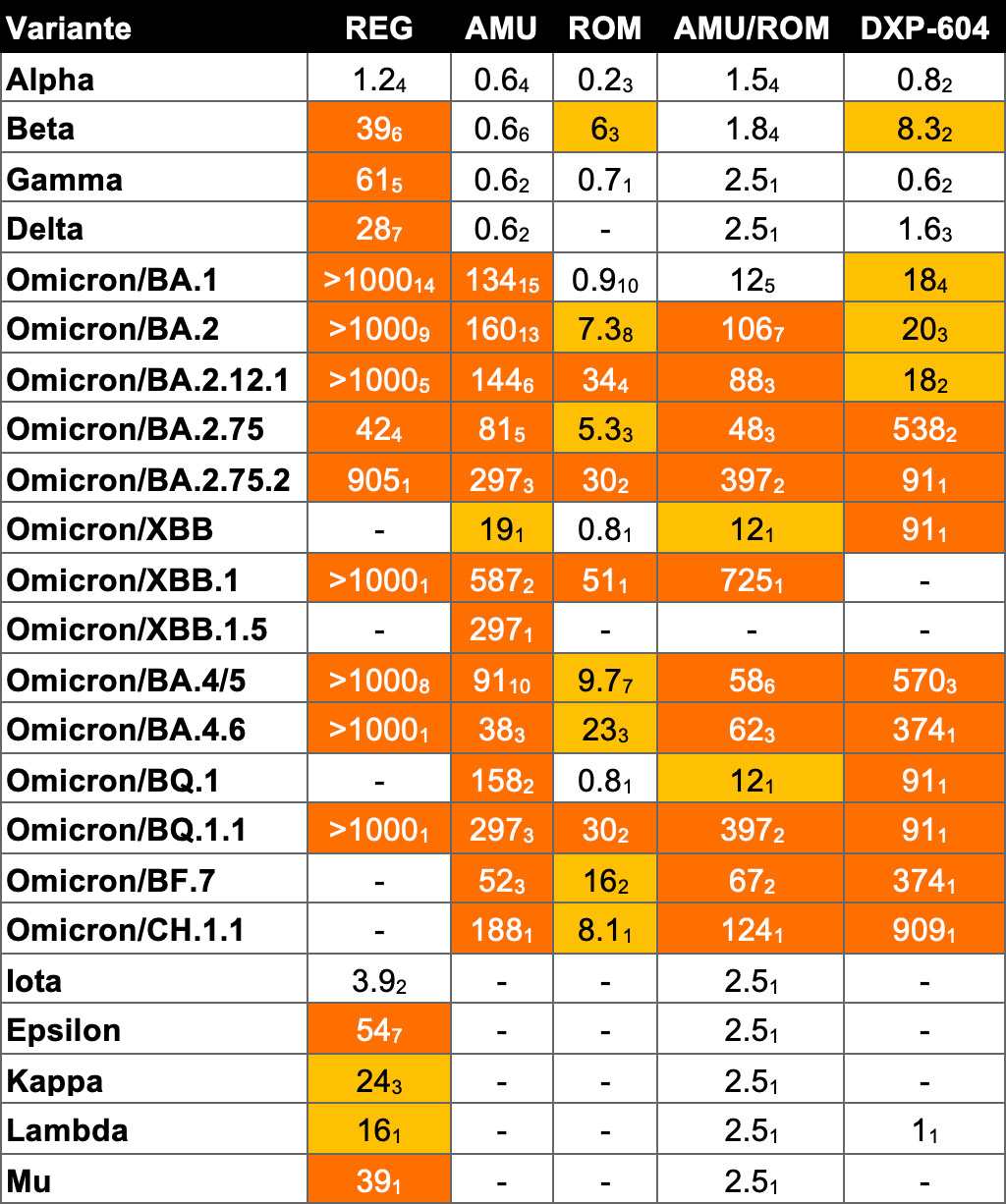
Tabla 1 (continuación)
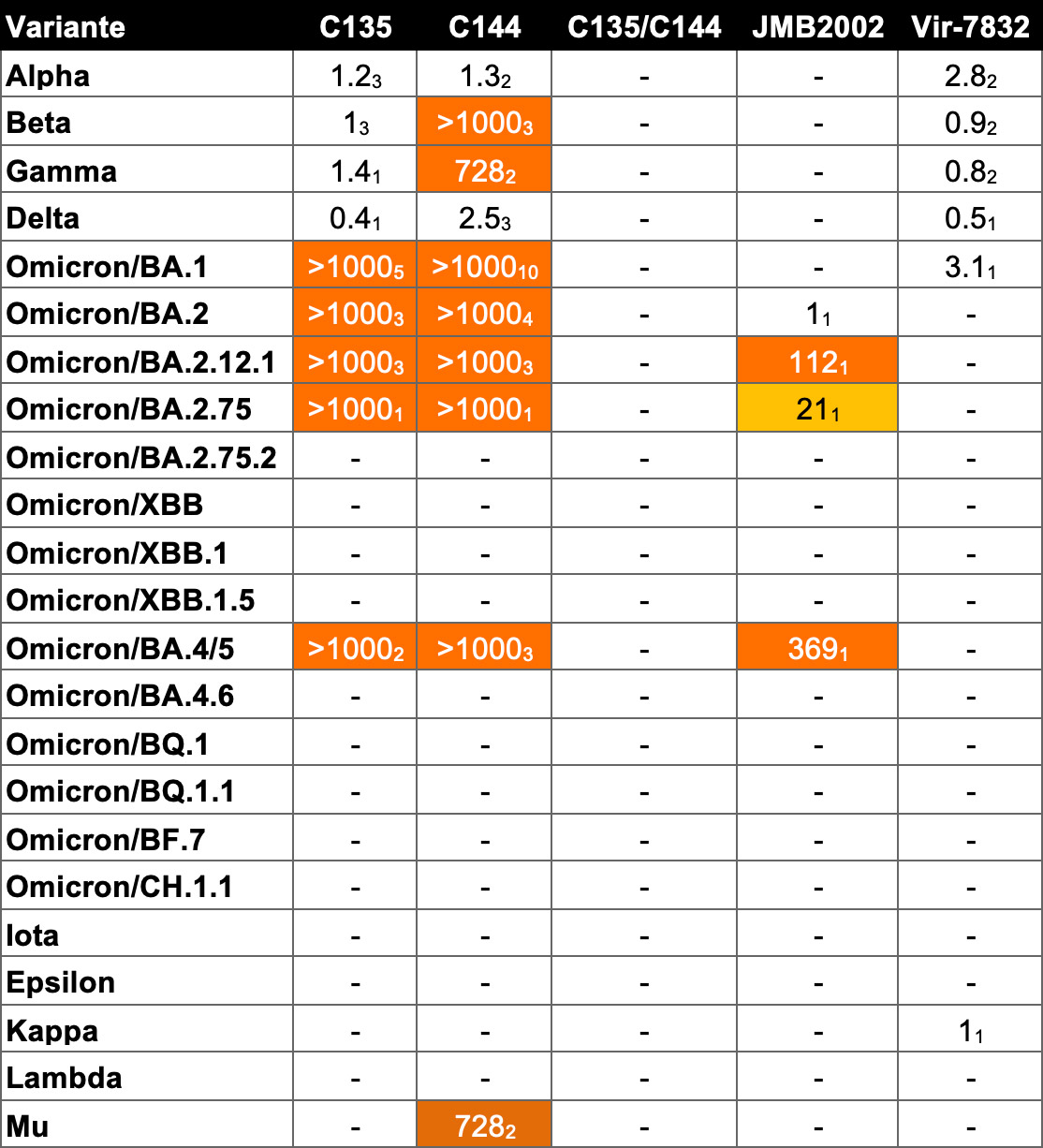
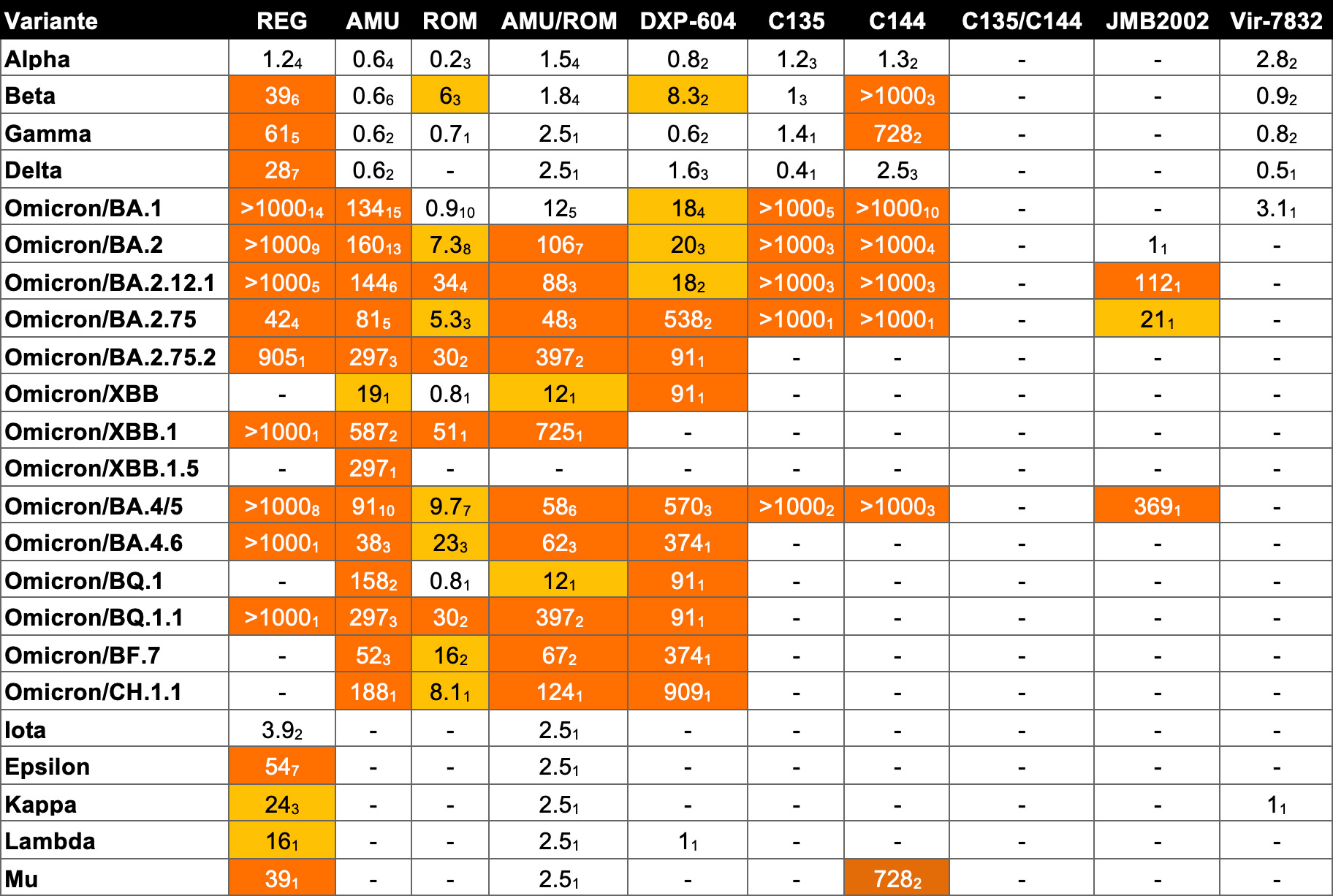
El esquema de colores indica la reducción en la neutralización:
- ausencia de color: susceptibilidad reducida <5 veces
- naranja claro: susceptibilidad reducida de 5 a 24,9 veces
- naranja oscuro: susceptibilidad reducida ≥25 veces
Fuente: Stanford University Coronavirus Antiviral & Resistance Database
• El valor de fold change es la mediana del valor de las muestras, el subíndice indica el número de muestras. – : ausencia de datos de susceptibilidad. * : conflicto entre datos de grupos de investigación.
• Abreviaturas de los anticuerpos monoclonales (mAb): BAM: Bamlanivimab/LY-CoV555; CAS: Casirivimab / REGN10933; IMD: Imdevimab / REGN10987; CAS/IMD: Casirivimab+imdevimab / REGN-COV2; ETE: Etesevimab/LY-CoV016/JS016/CB6; CIL: Cilgavimab/COV2-2130/AZD1061; TIX: Tixagevimab / COV2-2196 / AZD8895; TIX/CIL: Tixagevimab+Cilgavimab; BAM/ETE: Bamlanivimab+Etesevimab; SOT: Sotrovimab / Vir-7831 / S309; REG: Regdanvimab / CT-P59.
Actualizado: 10 marzo 2023